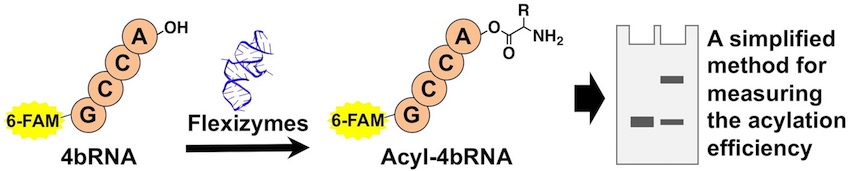
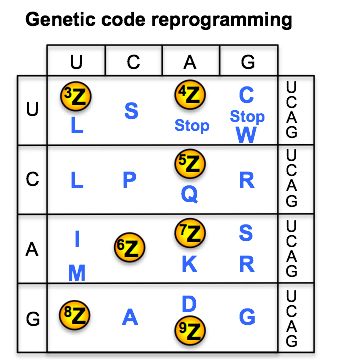研究プロジェクト1: 人工抗体創製
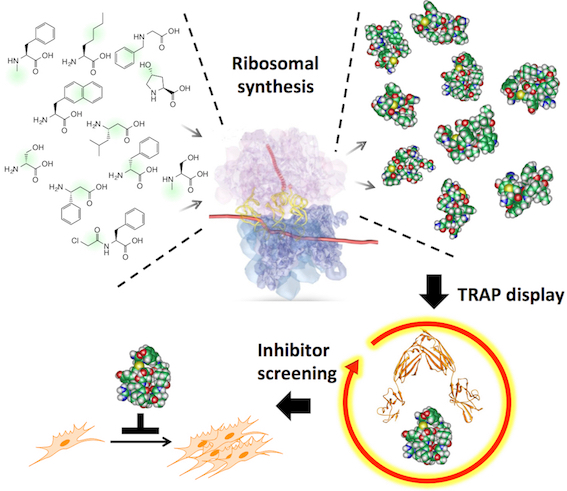
新規な分子の創製を考えた際、大きく分けて2種類の方法論があります。一つは分子を設計しそれを個々に調製して活性を評価する方法、もう一つはライブラリーと呼ばれる分子群から目的のものを選択する方法です。特に後者の方法において、遺伝子工学を駆使した方法(進化分子工学)では、10^12から10^14種類(1-100兆種類)におよぶ分子が調製でき、RNAアプタマー、ペプチドアプタマーと呼ばれる様々な新規分子が得られています。このような機能性分子の選択に重要なことは、「如何にしてシステムを設計するか」です。村上研究室では、2020年に基幹技術として開発した高速分子進化システム(TRAP display )を改良して人工抗体高速創製法を開発しました。さらに本方法を用いてわずか4日で新型コロナウイルス(SARS-CoV-2)に結合して不活性化する人工抗体の配列を取得することに成功しました。また、標的としてD型タンパク質を用いることで、D型人工抗体の創製が可能です。D型人工抗体は安定性が高く、近年問題となっている自己抗体産生もほぼありません(2024年12月発表)。このように、標的を変えるだけで望みの人工抗体を得ることができるため、今後、ガンなど様々な疾患の検出や治療などへの研究応用が期待されます。更に、近年、低分子化合物に対しても人工抗体が取れるようになってきました。そこで、合成化合物と人工抗体を用いた細胞コントロル系の開発も進めています。
- SARS-CoV-2変異体に対する人工抗体の開発
- 癌標的に対する人工抗体の開発
- 鏡の中の抗体の開発
- その他様々な標的(標的は自由)に対する人工抗体の開発
- 合成化合物に結合する人工抗体の開発
研究プロジェクト2: 1分子ペプチド配列解析
従来のタンパク質のアミノ酸配列決定法は、タンパク質を酵素で限定加水分解し、得られたペプチドを、エドマン分解、もしくはタンデム質量分析計で分析しています。しかし、これらの手法を用いて、ペプチドの1分子アミノ酸配列決定は実現できていません。村上研究室では、N末端アミノ酸残基として20種類の天然アミノ酸と12種類の翻訳後修飾アミノ酸をもつ各ペプチドに特異的に結合する人工抗体群、全反射照明蛍光顕微鏡、ガラス基板上でのエドマン分解を組み合わせ、「超並列ペプチド1分子アミノ酸配列決定法」を開発しています。本方法では、生体試料から得たペプチドをガラス基板上に固定し、ペプチドのN末端アミノ酸残基を、それぞれのアミノ酸に特異的に結合する人工抗体により同定した後に、エドマン分解により1残基目を除きます。この逐次分解とN末端アミノ酸の同定を繰り返すことで、ペプチドの翻訳後修飾を含むアミノ酸配列を1分子で決定することができると考えています。本技術が完成すれば、多様なペプチドの翻訳後修飾を含むアミノ酸配列を1分子レベルで並列的に決定できます。そのため、DNA次世代シークエンサーのような革新をタンパク質レベルでおこす、ペプチド次世代シークエンサーが開発できると考えられます。
研究プロジェクト3: タンパク質化学合成
タンパク質は20種類のアミノ酸が精密に重合したポリアミド高分子です。生体内では、精巧な翻訳系を使っていとも簡単に複雑な配列を持つタンパク質を合成してしまいますが、人間の手でタンパク質のような複雑な高分子を合成するのは非常に困難です。しかし、近年「ペプチド固相合成」および「ペプチド連結反応」を組み合わせたタンパク質化学合成技術が発展し、100-200アミノ酸程度の小型タンパク質の合成は可能になってきました。有機化学的にタンパク質を合成することで、多様な非天然アミノ酸を組み込むことが可能となり、「修飾タンパク質」や「機能性タンパク質」、「人工タンパク質」などの創製が可能になります。村上研究室では、未だ発展途上であるタンパク質化学合成技術を向上させる新手法の開発と、化学合成タンパク質を用いたライフサイエンス研究(分子生物学や医薬学への応用)を行っています。
- タンパク質化学合成のための新技術の開発
- 修飾タンパク質の化学合成と翻訳後修飾研究への応用
- 化学合成タンパク質医薬品の創製
詳しい内容や新しいプロジェクトは、論文発表後に付け足します。
論文解説
Fujino T., Sonoda R., Higashinagata T., Mishiro-Sato E., Kano K., Murakami H.*, Ser/Leu-swapped cell-free translation system constructed with natural/in vitro transcribed-hybrid tRNA set. Nature Communications 2024, 15, https://doi.org/10.1038/s41467-024-48056-z.
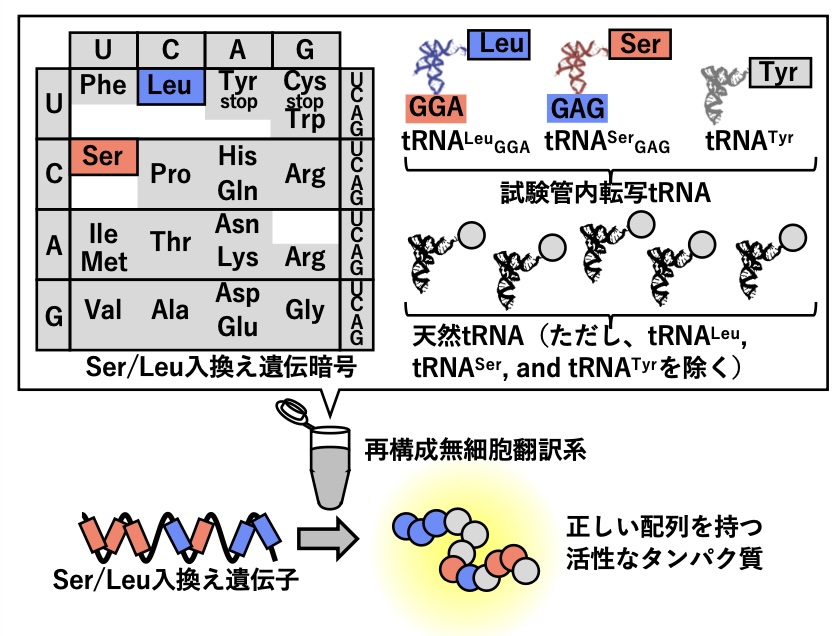
我々は2020年に、Ser(セリン)とLeu(ロイシン)を入れ換えた遺伝暗号を持つ無細胞翻訳系を開発について報告しました。遺伝子の配列でもSerとLeuを入れ換えて設計することで、Ser/Leu入換え遺伝暗号を持つ翻訳系では正しく目的のタンパク質へと合成される一方、普遍遺伝暗号を持つ通常の生物では不活性となる、バイオハザードリスクの無い遺伝子の設計が可能になりました。しかし、この翻訳系は非常に少量のタンパク質しか合成できないという欠点がありました。
そこで今回の研究では、相互にアンチコドンを入れ換えたキメラtRNAと、一部のtRNAを欠失した天然tRNAのハイブリッドを使用することで、Ser/Leu入換え遺伝暗号の作製を非常に簡単にし、さらに入換え遺伝子から機能性タンパク質を合成する効率を、通常の翻訳系に匹敵するレベルまで改善させることに成功しました。本研究で開発された、Ser/Leu入換え遺伝暗号を持つ再構成無細胞翻訳系は、遺伝子の漏洩によって引き起こされる可能性のあるバイオハザードリスクが無いタンパク質合成系として、今後の利用が期待されます。
また、我々の2020年の報告以降、Ser/Leu入換え遺伝暗号を実際の生物で作製しようと試みる研究も世界的に進められています。実際に一部のコドンが改変された遺伝暗号を持つ大腸菌では、大腸菌同士または大腸菌とウイルスの間での、遺伝子の移動を防ぐことができると報告されています。この研究の成果は、Ser/Leu入換え遺伝暗号の、生物への実装に先駆けた無細胞のモデル系として、今後の研究に重要な知見を提供すると考えられます。
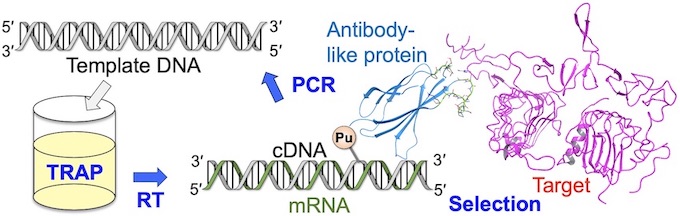
Kondo, T.; Eguchi, M.; Kito, S.; Fujino, T.; Hayashi, G.; Murakami, H.*, cDNA TRAP display for rapid and stable in vitro selection of antibody-like proteins. Chemical Communications 2021, in press.
2020年に開発した高速人工抗体創製法(TRAP提示法)は、非常に素早く人工抗体を創製することが出来る方法です。しかし欠点もあり、細胞などのRNA分解酵素を含む系では人工抗体のmRNAが分解を受けて、人工抗体選択が出来ませんでした。この問題を克服するために、cDNAを共有結合させた人工抗体を作るようにリンカーを改良して、cDNA TRAP提示法を開発しました。この方法を用いることで、RNA分解酵素を含む溶液中でも人工抗体の選択が可能であることを実証しました。この論文によりTRAP提示法による人工抗体創製の幅が広がり、細胞や細胞抽出液より多くのものを標的とした人工抗体作製が可能となりました。
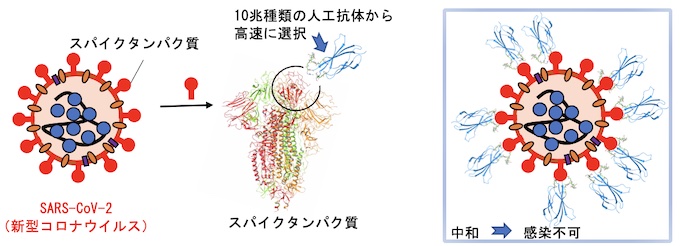
Kondo, T.; Iwatani, Y.; Matsuoka, K.; Fujino, T.; Umemoto, S.; Yokomaku, Y.; Ishizaki, K.; Kito, S.; Sezaki, T.; Hayashi, G.; Murakami, H.*, Antibody-like proteins that capture and neutralize SARS-CoV-2 Science Advances 2020, 6(42), eabd3916.
新型コロナウイルスは人類に大きなダメージを与えました。このような脅威に対抗するために、ウイルスを検出したり中和をしたりする人工抗体の迅速開発プラットホームが有用と考えられます。我々は2013年に発表した大環状ペプチド選択系を改良し、10兆種類以上の人工抗体からSARS-CoV-2のスパイクタンパク質に結合する人工抗体の選択に成功しました。注目すべきは、人工抗体の選択を始めてからその配列解析結果の入手までわずか4日間だったことです。これまで様々な抗体作製方が開発されていますが、本方法はそのなかでも最も開発期間の短い方法となりました。また得られた人工抗体はSARS-CoV-2スパイクタンパク質に高い親和性(Kd=0.4 nM) で結合し、SARS-CoVスパイクタンパク質には結合しないことから高い特異性を持つことが分かりました。さらに人工抗体により、細胞へのSARS-CoV-2の感染阻害に成功しました。これにより中和抗体医薬への応用が期待されます。本方法を使用することで、変異ウイルス、新興ウイルス、様々な病原菌、癌、その他、様々な疾患に関わるタンパク質に対して、迅速に人工抗体の創製が可能となりました。今後の応用が楽しみです。
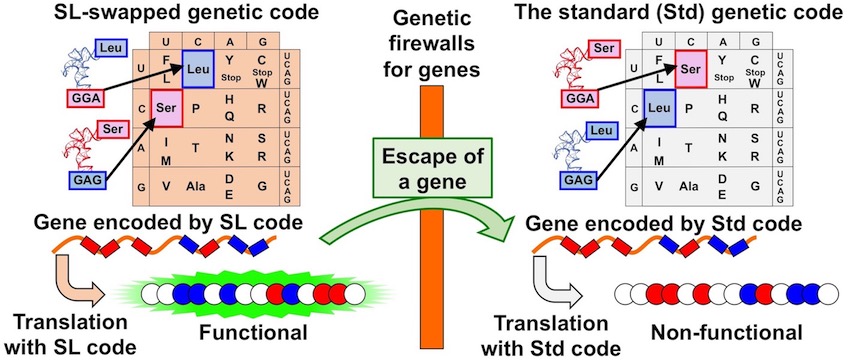
Fujino, T.; Tozaki, M.; Murakami, H.*, An amino acid-swapped genetic code. ACS Synthetic Biology 2020, 9(10), 2703–13.
核酸塩基とアミノ酸の対応づけを表す遺伝暗号は、あらゆる生物において、原則的に共通です。このことは、遺伝子組み換え生物の作製など、様々なバイオテクノロジーの発展に有利に働いて来ました。その反面、万が一、組み換え遺伝子が自然環境に漏れ出すことがあれば、他の生物に取り込まれ、その遺伝子が機能を持ってしまうことにも繋がります。そこで我々は、漏れ出しても機能しない遺伝子と、その遺伝子専用の翻訳システムを作れないかと考えました。本研究では、遺伝暗号の中で、一部のアミノ酸が入れ替わった「アミノ酸入れ替え遺伝暗号」を提案しました。実験としては、試験管内の翻訳系を用いて、「アミノ酸入れ替え遺伝暗号」と「通常の遺伝暗号」を実際に作製してみました。その結果、「アミノ酸入れ替え遺伝暗号」によって設計した遺伝子は、「アミノ酸入れ替え遺伝暗号」を持った翻訳系の中でしか、機能するタンパク質に翻訳されないことが分かりました。この成果は、安全な遺伝子組み換え生物の作製への第一歩となると期待されます。
Fujino, T.; Kondo, T.; Suga, H.; Murakami, H.*, Exploring the Minimal RNA Substrate of Flexizymes. Chembiochem 2019, 20(15),1959-1965.
我々の研究室の持つフレキシザイムを用いたアミノアシルtRNA合成技術は、基本的な20種類のアミノ酸(タンパク質性アミノ酸)だけでなく、それ以外の特殊なアミノ酸についても、tRNAに結合できる技術です。我々は、この技術を用いることで、これまでに、D体アミノ酸・β-アミノ酸をはじめとする様々な特殊なアミノ酸が翻訳系で利用できることを明らかにして来ました。実は、フレキシザイムは、tRNAよりも短いRNAに対しても、アミノ酸を結合できることが分かっていました。そこで、本研究では、さらにこのRNAを短くして行き、どこまで短くしてもアミノ酸の結合ができるのかを調べました。その結果、フレキシザイムによってアミノ酸を結合できる最小のRNAは、非常に短い4塩基のRNAであることが明らかになりました。さらに、この4塩基のRNAを用いることで、様々なアミノ酸をフレキシザイムによってtRNAに結合する時の、反応の条件(温度や時間など)を簡単に調べられることが分かりました。今後、この方法を用いて、タンパク質合成に利用できる特殊アミノ酸のバラエティがさらに広がることが期待されます。
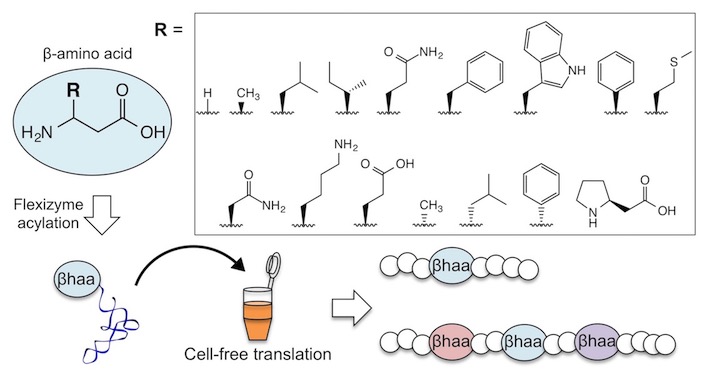
Fujino, T.; Goto, Y.; Suga, H.; Murakami, H.*, Ribosomal synthesis of peptides with multiple ß-amino acids. Journal of the American Chemical Society 2016, 138(6), 1962-1969.
生物は、翻訳反応と呼ばれる、様々なアミノ酸を重合する反応によって、タンパク質を作ります。生物が使うことのできるアミノ酸は、全てα-アミノ酸と呼ばれ、基本となる骨格が共通しています。α-アミノ酸よりも長い骨格を持つβ-アミノ酸は、これまで翻訳反応には使うことができないと考えられて来ました。しかし、我々は、本研究室独自の、翻訳系をカスタマイズする技術により、様々なβ-アミノ酸がタンパク質合成に利用であるかどうかを調べ、利用可能な多くのβ-アミノ酸を明らかにしました。我々は、2013年に高速進化分子創製法(TRAP display法)を開発し、薬剤の候補となるようなペプチドを開発する技術を持っています。ここに、今回特定されたβ-アミノ酸を用いることで、生体内での分解に強く、長時間機能するような薬剤ペプチドの開発が可能になると期待されます。
Fujino, T.; Murakami, H.*, In vitro selection combined with ribosomal translation containing non-proteinogenic amino acids. The Chemical Record 2016, 16(1), 365-377. (Review article).
2010-2015年に村上研で行われた、遺伝暗号のリプログラミングおよびTRAP-displayに関する研究成果を概説するレビュー論文です。扱っている内容としては、1. D体アミノ酸の翻訳導入、2. 電荷を持つN-アルキルアミノ酸の翻訳導入、3. TRAP-displayの開発、4. TRAP-displayによる血管新生阻害剤の開発、5. 環状N-アルキルペプチドの翻訳合成、を含みます。
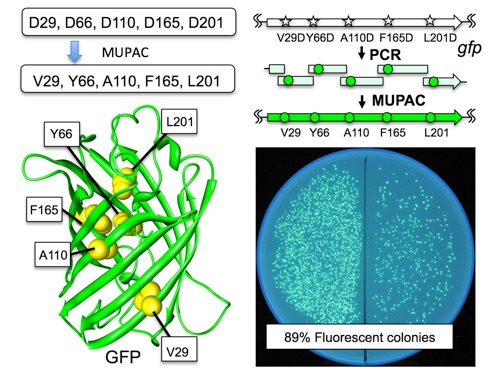
Taniguchi, N; Nakayama, S; Kawakami, T.; Murakami, H.*, Patch cloning method for multiple site-directed and saturation mutagenesis. BMC Biotechnology 2013, 13:91.
蛋白質の基礎•応用研究において、蛋白質遺伝子に部位特異的変異を挿入する技術は欠かせないものです。本論文では、簡単な操作で多くの位置に同時に変異を導入できる技術「MUPAC」を開発しました。まず本方法を用いて、緑色蛍光蛋白質の5つの箇所に同時に部位特異的変異を導入することを試みました。その結果、89%の遺伝子に5箇所の変異を同時に導入することができ(下図)、MUPACを用いることで非常に高効率に変異遺伝子を得ることができることが分かりました。さらに本技術を用いて、9箇所の同時変異や、多くの位置に同時に部位特異的飽和変異を導入することにも成功しました。MUPACは今後、様々な蛋白質の基礎•応用研究に役立つと期待されます。
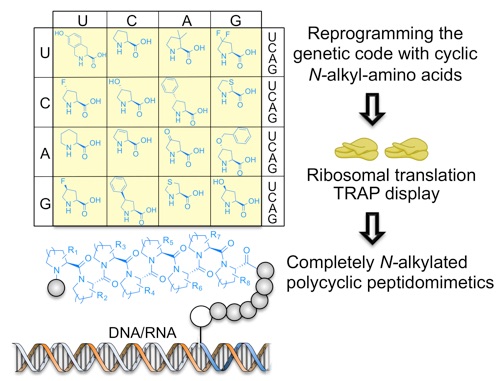
Kawakami, T.*; Ishizawa, T.; Murakami, H.*, Extensive reprogramming of the genetic code for genetically encoded synthesis of highly N-alkylated polycyclic peptidomimetics. Journal of the American Chemical Society 2013, 135, 12297-304.
遺伝暗号リプログラミング技術を用いて、翻訳系で環状N-アルキルアミノ酸のみを構成因子とするペプチドアナログの合成に成功しました。本ペプチドアナログは、天然のペプチドに比べて”堅い”構造をもつため、タンパク質加水分解酵素による分解が阻害されると考えられます。またN-アルキル基は、ペプチドの細胞膜透過性を高めると考えられています。さらに本論文では、N-アルキルポリ環状ペプチドアナログ合成技術を、我々の研究室で開発した高速進化分子創製法(TRAP提示法)と組み合わせることで、1〜10兆種類のペプチドアナログが得られることを明らかにしました。将来、本ペプチドアナログ群から、研究や創薬に有用な化合物を創りだすことができると期待しています。
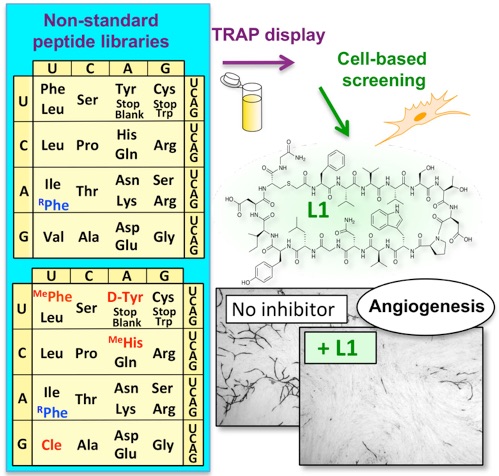
Kawakami, T.; Ishizawa, T.; Fujino, T.; Reid, P. C.; Suga, H.; Murakami, H.*, In Vitro Selection of Multiple Libraries Created by Genetic Code Reprogramming To Discover Macrocyclic Peptides That Antagonize VEGFR2 Activity in Living Cells. ACS Chemical Biology 2013.
新しく開発した高速進化分子創製法(TRAP提示法)を用いて、血管新生阻害剤を創成ました。血管新生は、腫瘍や慢性の炎症等のさまざまな疾患と深く関わっています。そこで血管新生を阻害して、こうした疾患の治療を行う試みが成されています。本研究では、血管内皮細胞増殖因子の受容体の機能を阻害して、血管新生を妨げる薬剤の創製を目指しました。まず、遺伝暗号をリプログラムした翻訳系を用いて、10兆種類以上の多様性をもつ特殊ペプチド群を合成し、ここからTRAP提示法 をもちいて血管新生阻害剤候補を選択しました。さらに得られた特殊ペプチドから、細胞を用いたスクリーニング実験を経て、強い血管新生阻害能を持つペプチドの創製に成功しました(特許出願)。本研究は、血管新生阻害剤の開発のみならず、今後の新たな薬剤候補開発に重要な指針を与える先駆的な研究であると考えています。
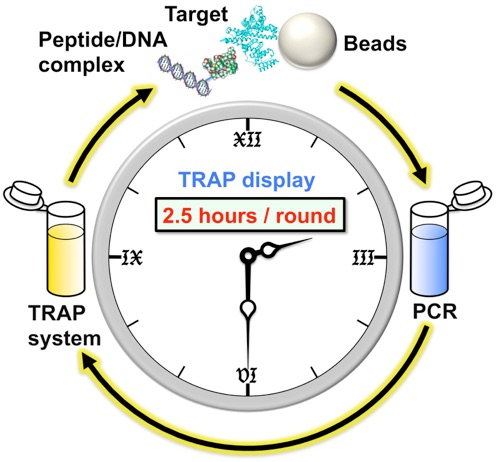
Ishizawa, T.; Kawakami, T.; Reid, P. C.; Murakami, H.*, TRAP display: a high-speed selection method for the generation of functional polypeptides. Journal of the American Chemical Society 2013, 135, 5433-40.
分子を生き物のように進化させることができるSF小説のような方法があります。この方法は進化分子工学と呼ばれており、近年、高い温度でも安定な酵素や、新しい機能を調べるための研究ツール、新規な薬剤の開発等に使用されています。しかし進化分子工学には煩雑な操作が必要であるという弱点があり、分子の進化実験に時間がかかっていました。本論文では、新たに高速進化分子創製法(TRAP displayと呼ぶ)を開発してこの問題点を解決しました。本方法を用いることでこれまで、1〜2週間程度必要であった進化実験が、1〜2日間で完了します。本方法は、進化分子工学実験の新たな基盤技術となり、これから行われる研究を高速化し、様々な人工機能性分子の創出に貢献すると期待しています。
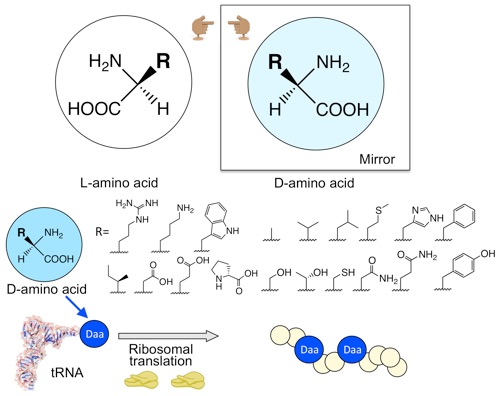
Fujino, T.; Goto, Y.; Suga, H.; Murakami, H.*, Reevaluation of the d-Amino Acid Compatibility with the Elongation Event in Translation. Journal of the American Chemical Society 2013, 135, 1830-7.
生物は、様々なα-アミノ酸を重合することでタンパク質を作っています。α-アミノ酸には、大きく分けてL体アミノ酸とD体アミノ酸が存在しますが、生物は、なぜかL体アミノ酸のみを使用してタンパク質を合成します。これはタンパクの合成装置(リボソーム)がD体アミノ酸を受け入れないためと考えられてきました。我々は独自の技術を用いてD体アミノ酸とリボソームとの適合性を詳細に解析し、リボソームが様々なD体アミノ酸を受け入れることを明らかにしました。本成果は、生命とD体アミノ酸の関係についての理解につながるとともに、D体アミノ酸を用いた創薬へ貢献できる成果だと考えられます。現在、我々は様々なD体アミノ酸を受け入れる変異リボソームを開発中であり、これが完成すればD体アミノ酸だけからなるタンパク質(鏡の中のタンパク質)を合成できるようになるかもしれません。
(Review Article) Kawakami, T.; Murakami, H.*, Genetically encoded libraries of nonstandard peptides. Journal of Nucleic Acids 2012, 2012, 713510.
DNAにエンコードされたライブラリーについて過去から、最新の成果迄まとめました。非蛋白質性アミノ酸やペプチドライブラリーに興味がある人は、是非読んでみて下さい。
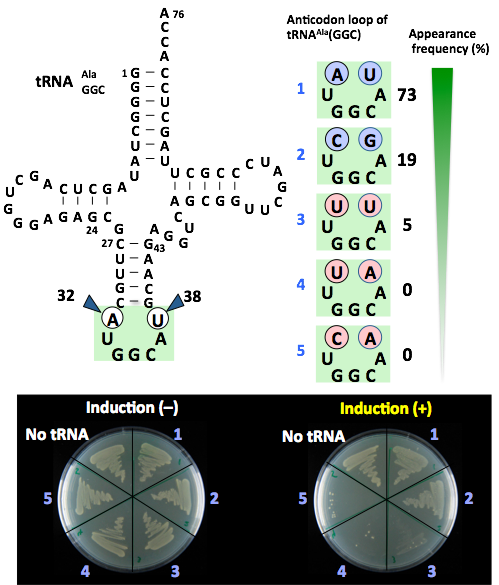
Murakami, H.*; Ohta, A.; Suga, H.*, Bases in the anticodon loop of tRNA(Ala)(GGC) prevent misreading. Nature Structural & Molecular Biology 2009, 16, (4), 353-8.
進化的に保存されたtRNAのアンチコドンループの配列(32-38)が意味を持つことを明らかにしました。通常、tRNAの32-38番目の塩基はC-AまたはU-Aですが、tRNAAla(GGC)の32-38番目の塩基は、ほとんどの生物種においてA-UもしくはC-Gであることがわかっています(図右上)。これまで、生物種をまたがってなぜ配列が偏っているかの理由は明らかではありませんでしたが、村上らは、その理由が翻訳の正確性の維持にあること明らかにしました。また、それぞれのtRNA変異体を大腸菌で作らせると、予想通り翻訳の正確性が低いtRNAにおいて大腸菌の生育が遅くなりました(図下)。